2019年6月15日,《Horticulture Research》杂志以Article形式在线发表了海南大学林学院热带特色花木资源团队研究论文“MDR: an integrative DNA N6-methyladenine and N4-methylcytosine modification database for Rosaceae”。《Horticulture Research》是Nature出版集团旗下唯一园艺领域顶级期刊(一区,IF=3.37)。海南大学为该论文第一完成单位。

DNA甲基化是表观遗传调控的重要组成部分,在生物发育及代谢中发挥着重要作用。近年来,新兴的第三代测序技术为全基因组碱基水平检测DNA甲基化提供重要的技术支撑。基于三代测序的DNA甲基化已在人、果蝇、水稻、拟南芥等模式生物中被成功解析,成果多见于CNS或其子刊,已然成为表观遗传学研究领域的热点。然而园艺植物DNA甲基化解析鲜有报道,尤其是近年刚被报道在真核中存在的DNA-6mA和DNA-4mC两种甲基化类型。
蔷薇科作为园艺植物的一个重要科属,包含了2500多个种,具有重要的观赏和食用价值等价值。该项研究收集了目前所有已释放的蔷薇科三代测序数据,首次在蔷薇科植物中破译DNA-6mA和DNA-4mC修饰图谱,揭示了其在基因组功能区中的分布特征,并整合蛋白编码基因信息探析其在基因表达中的作用。主要工作包括了蔷薇科林地草莓(Fragaria vesca)和月季(Rosa chinensis)在染色体、样本和基因三个水平的DNA-6mA和4mA的甲基化差异,以及甲基化位点保守性Motif和GO功能富集分析(图1)。为了将所研究结果更好地展现和服务于相关领域的科研工作人员,该论文基于DNA甲基化分析结果,开发了首个蔷薇科6mA和4mC的在线数据库MDR(mdr.xislab.org)。MDR提供全基因组DNA-6mA和DNA-4mC甲基化信息的存储、浏览、搜索和比较功能(图2),实现DNA甲基化位点的在线可视化和表达分析。随着越来越多的蔷薇科三代测序产生,MDR数据库也将不断更新和完善,有望为园艺植物蔷薇科表观遗传学研究发挥积极作用。
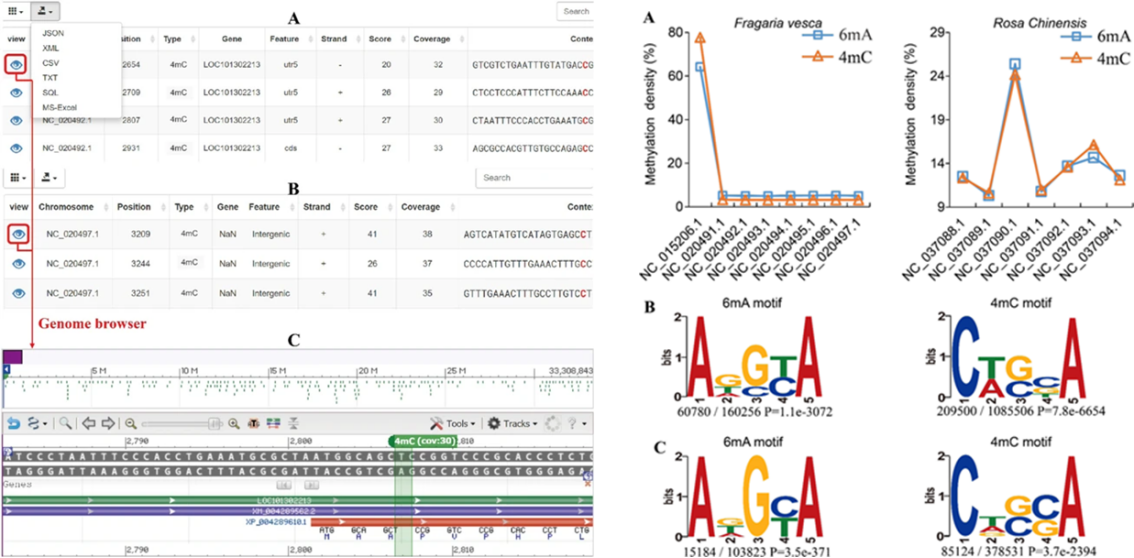
图1.蔷薇科植物中DNA-6mA和4mC的分布特征与保守序列Motif

图2. MDR数据库的功能与内容
林学院热带特色花木资源团队成员刘照宇(本科生),邢剑锋,陈伟,栾美薇参与完成了该项研究工作,其中刘照宇,邢剑锋,陈伟为共同第一作者;海南大学林学院谢尚潜博士为该研究第一通讯作者,中山大学肖传乐副研究员为共同通讯作者。
林学院热带特色花木资源团队是林学院热带特色林木花卉遗传与种质创新教育部重点实验室、海南省热带特色花木资源生物学重点实验室的核心团队之一。谢尚潜博士于2017年5月以海南大学“D类”人才引进,目前作为该团队的骨干成员,已经以海南大学为署名单位在Nature Methods,Horticulture Research,Journal of Proteomics,BMC genomics等专业领域杂志上发表SCI论文7篇,其中第一署名单位4篇(一区1篇,二区2篇以及三区1篇)。
论文链接:https://www.nature.com/articles/s41438-019-0160-4
 海南大学
海南大学