近日,国际学术期刊《Computational and Structural Biotechnology Journal》(JCR一区,IF=7.271)在线发表了以海南大学热带特色林木花卉遗传与种质创新教育部重点实验室为第一单位,题目为“SCSit: A high-efficiency preprocessing tool for single-cell sequencing data from SPLiT-seq”的学术论文。
细胞异质性是生物学中普遍存在的一种现象,近年发展起来的高通量单细胞测序技术为单细胞水平鉴定细胞类型提供了可能。相较于组织水平容易忽略细胞特异性表达的问题,单细胞测序具有明显优势。单细胞SPLiT-seq测序技术利用四轮组合条形码(Barcodes)和唯一分子标识符(UMI)标记转录组数据的细胞来源(图1)。基于Barcodes的索引信息,SPLiT-seq技术无需对单个细胞进行分离,足以特异性地标记超过100万个细胞。这一原理也极大降低了单细胞测序的成本和实验要求,使其在单细胞研究中得到广泛应用。然而,目前对于SPLiT-seq数据缺乏专门的自动化和快速的处理工具,现有的方法简单地基于常规比对工具,如BLAST或BWA,对于同时识别不同区域的所有三个Barcodes计算相对费时。另外,也缺乏专门针对Barcodes数据的容错匹配自动标注方法,以致SPLiT-seq测序下机数据预处理后的可使用率有限。
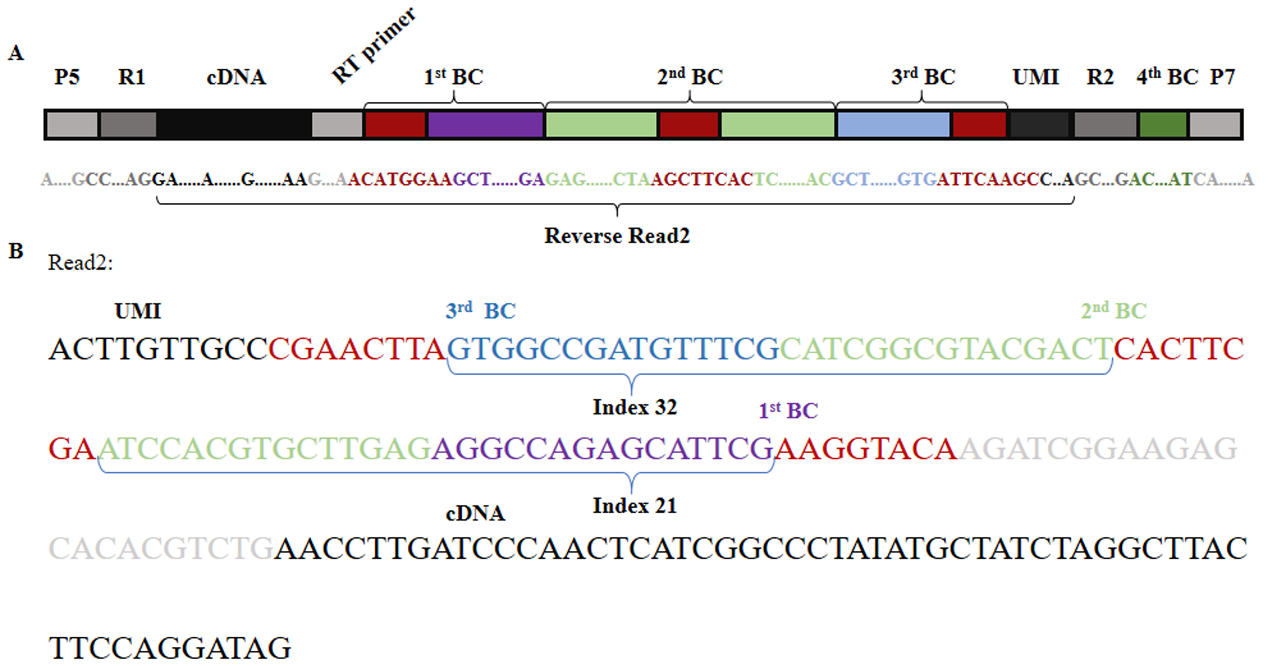
图1. SPLiT-seq数据的条形码示意图
该论文针对上述SPLiT-seq单细胞测序数据处理存在的问题,开发了一种高效SPLiT-seq单细胞测序数据的预处理工具SCSit(https://github.com/shang-qian/SCSit),SCSit可直接快速识别并标记Barcodes和UMI组合以实现自动预处理和单细胞分类,另通过在比对过程中准确识别插入和缺失以显著增加下机数据的使用率。利用人和鼠细胞混合的5个样本的测序数据比较SCSit新方法和传统分步数据处理方法,结果表明SCSit识别的测序数据(reads)与传统方法相比一致率达97%,处理后可识别的reads数目是传统方法的两倍,且运行时间仅是传统方法的10%。该工具有效提高了SPLiT-seq单细胞测序平台的数据处理效率。总的来说,该工具实现了自动识别Barcodes和UMI,并在比对中可准确检测数据的插入和删除(图2),显著增加了测序数据预处理后的产出率,从而实现了高效的SPLiT-seq单细胞数据的分类和利用。

图2. 测序数据的Index容错匹配示意图
该论文以海南大学为第一和通讯作者单位,热带特色林木花卉遗传与种质创新教育部重点实验室研究生栾美薇和王也凡参与了该项研究工作。2018级博士研究生栾美薇和海南医学院生物医学信息与工程学院林加论为该研究共同第一作者,海南大学林学院谢尚潜为通讯作者。该工作得到国家自然科学基金(31760316,32060149和31871326)、海南省自然科学基金(320RC500和ZDKJ201815)以及海南大学科研启动基金(KYQD(ZR)1721)等项目的支持。
论文链接:https://doi.org/10.1016/j.csbj.2021.08.021。
 海南大学
海南大学